MicroMón es un proyecto de aprendizaje servicio internacional entre ciertas universidades y centros de educación secundaria obligatoria y bachillerato que pretende motivar y hacer crecer el interés de los estudiantes en el mundo de las ciencias experimentales y, además, abordar uno de los problemas más presentes e importantes para la salud humana: la resistencia a los antibióticos y la consiguiente disponibilidad reducida de antibióticos eficaces frente a infecciones bacterianas.
Los antibióticos permiten tratar infecciones en los seres humanos, animales y plantas, ya que son “armas químicas” que eliminan a los microorganismos. Sin embargo, la naturaleza es quien los produce, pero también quien les otorga unos mecanismos de resistencia que convertirán a las bacterias patógenas en resistentes, de forma que si estos medicamentos ya no reaccionan correctamente contra las infecciones bacterianas, vencerlas será una tarea casi imposible.
De este modo, uno de los objetivos de este proyecto de investigación es encontrar si, a partir de una muestra de suelo, algún microorganismo aislado puede producir sustancias antibióticas que inhiban el crecimiento de bacterias patógenas, para que posteriormente los centros de investigación, como la Universidad de Valencia, en nuestro caso, pueda identificarlas posteriormente con más detenimiento. Para realizarlo, el proyecto se ha dividido en cinco sesiones, en las que hemos participado plenamente los alumnos e, incluso, miembros de la misma Universidad:
En clase, en primer lugar, nos organizamos en parejas, después de presentar los objetivos y en qué consistía el proyecto, nos repartieron una bolsa sellada con una hoja de datos y diferente material estéril. En el tubo de plástico, también estéril, recogimos nuestro gramo de muestra (era importante encontrar un suelo inexplorado, de acceso público, puesto que había más posibilidad de conseguir microorganismos). Nuestra muestra corresponde al código V-ONT-12: fue recolectada en una sierra de nuestro pueblo cercana al Camino de la Solana, con un terreno húmedo y rocoso.

Para cultivar los microorganismos del suelo utilizamos los medios de cultivo TSA sencillos con pH neutro, por su facilidad a la hora de aislar bacterias. En la sesión 2 el primer paso a seguir fue rotular cinco tubos falcon de 15ml (10-1, 10-2, 10-3, 10-4, 10-5) y tres placas de Petri con agar (10-3, 10- 4, 10-5) con nuestro código y la disolución específica, y empezar a trabajar con el gramo de muestra. El objetivo era realizar cuatro disoluciones seriadas añadiendo la muestra en el primer tubo, agitándolo durante 30 segundos. Del resultado extrajimos un 1 ml para transferirlo al siguiente tubo, repitiendo el proceso con todos ellos por igual. Por último, tomamos dos gotas de disolución y las añadimos a las placas, empezando por la más diluida, porque en la siguiente sesión pudimos detectar diferentes colonias visibles en la superficie.
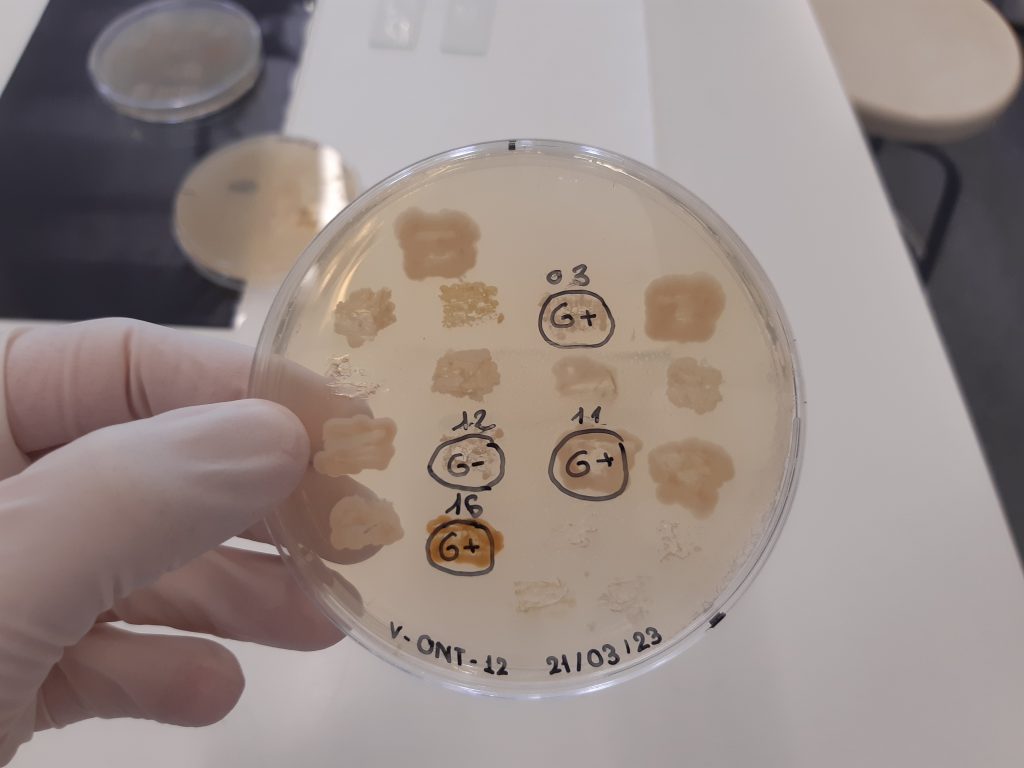
En la tercera sesión observamos macroscópicamente la morfología, tamaño y aspecto de las colonias que habían aparecido. Debíamos elegir entre las tres placas una donde se apreciaron y se pudieron aislar con mayor facilidad estas colonias, de modo que nosotros elegimos la placa 10-4, de la que diferenciamos 19 colonias de entre 140 diferentes, más o menos. Respecto a su forma, elevación y margen, la mayoría se caracterizaban por ser circulares, llanuras y con un margen entero o erosionado (se diferenciaban, sobre todo, por su color o transparencia). Al
identificarlas era el momento de subcultivarlas. Para llevarlo a cabo necesitábamos otra placa Petri; con ayuda de un palillo estéril, tocábamos la superficie de una colonia y la extendíamos en la nueva placa, sin pinchar y siguiendo el orden de una cuadrícula de referencia. (En esta sesión debíamos realizar la caracterización del suelo y en nuestra placa contábamos con 1,4 x 10e6 UFC/g de suelo).

En las próximas y últimas sesiones contamos con la ayuda de un profesor y distintos alumnos de la Universidad de Valencia. De todos los microorganismos aislados había que averiguar si alguno de ellos podía inhibir el crecimiento de bacterias patógenas en los seres humanos. Los patógenos que gastamos en el proyecto fueron Bacillus cereus, un organismo control Gram-positivo, y el Escherichia coli, un organismo control Gram-negativo, ya que existen antibióticos que son específicos para cada patógeno o para los dos a la vez y, por tanto, hay que comprobar su eficacia (se diferencian en que los Gram- poseen una bicapa lipídica muy impermeable para sustancias químicas, por lo que no pueden atravesarlas. En consecuencia, son más
difíciles de encontrar las que sí que lo hacen).
Por un lado, en la cuarta sesión, replicamos nuestra “placa maestra” transfiriendo cada colonia a
dos placas diferentes con una de las bacterias patógenas anteriores. Debíamos seguir el mismo orden que en la primera placa, utilizando también un palillo estéril por cada colonia y placa – nos recomendaron sembrarlas siguiendo la técnica de las tres estrías.
Por último, y tras la incubación de las nuevas placas, llegó el momento de observar si alrededor de los microorganismos habían aparecido zonas de inhibición de crecimiento del microorganismo testigo. La quinta sesión se basaba, por tanto, en la observación y comprobación de los resultados. Como bien nos explicaron, los resultados y la aparición de estas zonas dependían de la casualidad, pero nosotros tuvimos la oportunidad de apreciar estos halos en torno a microorganismos en las dos placas: cuatro zonas en el Gram+ y una en el Gram-.

Para determinar el tipo de pared celular de los aislados de interés, depositamos una gota de KOH sobre un portaobjetos y extendimos parte de la colonia problema sobre ella, mezclando durante 2 minutos. Si al levantar suavemente el palillo se formaba un filamento, la bacteria era Gram-, y si no se formaba, Gram+. Entre todas nuestras colonias, una de ellas dio positivo en Gram-, en específico la colonia 12, por lo que habíamos logrado encontrar un microorganismo positivo que la Universidad analizaría e identificaría posteriormente.
Por tanto, participando en este proyecto, hemos podido realizar diferentes experimentos con una sola muestra de suelo y, durante cinco sesiones trabajando con ella, observar los resultados obtenidos, en nuestro caso positivos, interpretar diferentes funciones microbianas utilizando, además, técnicas que nos permiten conocer una parte diferente de la biología.